單細胞分析可以研究細胞群內的細胞變異,是深入瞭解腫瘤異質性與疾病藥物開發的關鍵方法,而成功的單細胞基因組分析起始於合適的全基因組擴增 (WGA) 步驟,再搭配優化的定序方法和生物資訊分析來執行。
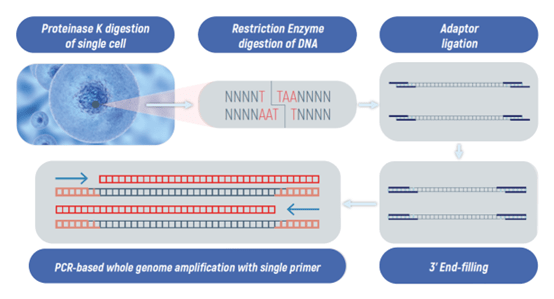
Ampli1 WGA是市面上唯一「非隨機」全基因組擴增方法
- 原理
- Ampli1 WGA採單一高特異性引子進行PCR,確保兩個等位基因平均擴增。
- 特點
- 平均且穩健的擴增技術,適合應用單細胞與少量細胞分析流程。
- 和其它擴增技術相比,提供更強大且高再現性的擴增效率。
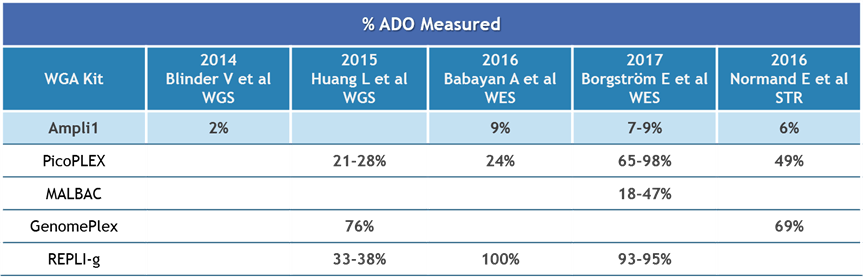
- 等位基因丟失率 (ADO) 最低,不再錯過重要的遺傳數據。
▲ 特定限制酶切位搭配單一引子進行PCR擴增,達到穩定且高再現性的擴增效果。
👉 瞭解更多 Ampli1 WGA PLUS Kit
等位基因丟失率低
- 大多數WGA方法採隨機擴增,容易造成兩個等位基因不平均放大,導致ADO比例高。
- Ampli1™ WGA特殊的設計原理可以提供穩定的擴增效率,減少放大不平衡的情況並降低ADO發生機率。
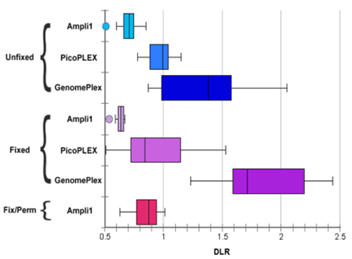
適用活細胞與固定細胞的強大擴增能力
- 產品適用性高,不需針對不同樣品購買不同試劑盒。
- 和其它WGA方法相比,Ampli1™ WGA擴增後在新鮮細胞與固定細胞的系統雜訊 (DLR) 都較低,對於後續進行拷貝數變異可取得更高解析度的分析結果。
與市面上八種WGA試劑盒進行多面向的擴增效率評估
2022年發表於International Journal of Molecular Sciences的文獻。
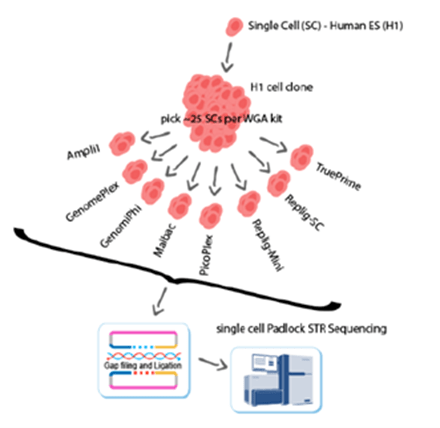 |
作者取已知的單一克隆細胞,利用八組不同品牌的WGA 試劑盒進行擴增,每組擴增 25 個單顆細胞,最後再通過 STR 評估 WGA 的擴增效果。 STR 對於重複單元的擴增錯誤非常敏感,可利用異型合子表現豐富的位點來評估 WGA 試劑盒的均勻擴增效果。 文章指出,使用 Ampli1 進行全基因組擴增可以最平均擴增等位基因且細胞間的再現性最高,是單細胞基因組異型合子比較分析的最佳選擇。 |
| 實驗結果 |
Positive control (文中未提及,應未經擴增)
◌ 取群細胞 Bulk作為控制組,與單細胞擴增結果作比較。
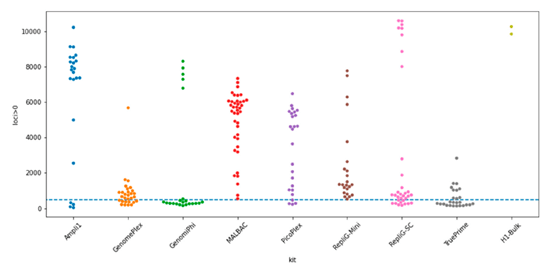
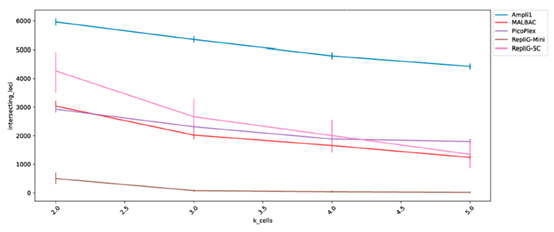
1) 單一細胞擴增覆蓋
-
- 每個點代表一個單細胞的擴增結果,位置越高表示單顆細胞被擴增的位點越多。
- 結果顯示Ampli1大部分的擴增結果落在較高的區域。
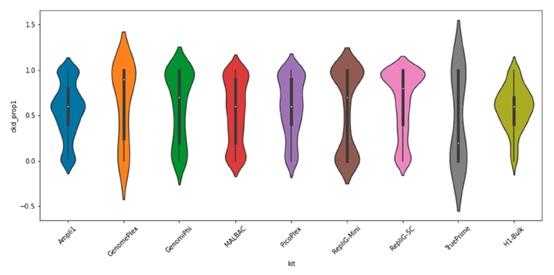
2) 異型合子等位基因平均擴增
-
- 趨近0.5表示完美平衡擴增結果,趨近0/1標示不均勻擴增。
- 結果顯示Ampli1大部分的擴增結果集中在0.5附近,僅次於群細胞的擴增結果。
3) 擴增結果的再現性
-
- 觀察在2~5個單細胞 (x-axis)中,重複擴增成功的位點 (y-axis)。
- 結果顯示Ampli1擴增的再現性遠高於其它品牌。
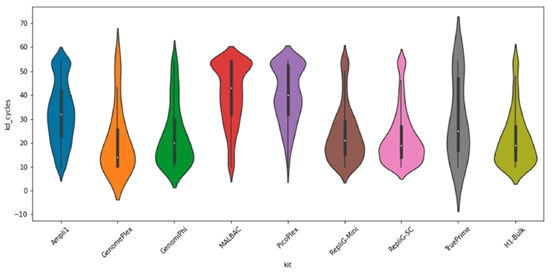
4) 擴增噪值
-
- 噪值分佈位置越低越好。
- 結果顯示在Ampli1、Malbac和PicoPlex觀察到較多雜訊,作者歸納原因來自擴增原理的差異: MDA試劑盒相使用的Phi29 DNA準確性較高且反應溫度較低,產生雜訊的機率較少。
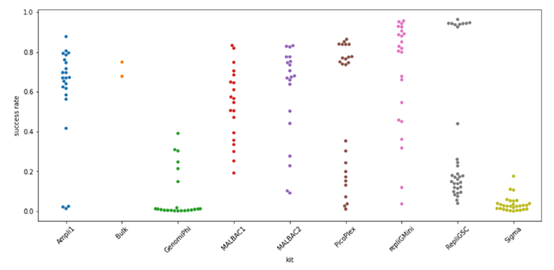
5) 成功率
-
- STR圖譜比較reference基因組,評估擴增成功率,位置越高表示成功率越高。
- 結果顯示Ampli1大部分的擴增結果落在較高的區域。
| 作者結論 |
全基因組擴增是單細胞研究的重要因素,作者比較八種市售WGA試劑盒,測量大量基因座與等位基因,觀察到Ampli1是最能平均放大等位基因的產品。
雖然從這次和過去的WGA比較可以發現目前並沒有萬能的WGA產品,但考量整體成功率與一致性,作者選擇Ampli1作為STR平台最合適的擴增試劑盒。
參考文獻
1. Binder, V. (2014). A New Workflow for Whole-Genome Sequencing of Single Human Cells. Human Mutation, 35(10), 1260–1270.
https://doi.org/10.1002/humu.22625
2. Huang, L. (2015). Single-Cell Whole-Genome Amplification and Sequencing: Methodology and Applications. Annual Review of Genomics and Human Genetics, 16(1), 79–102.
https://doi.org/10.1146/annurev-genom-090413-025352
3. Babayan, A. (2016). Comparative study of whole genome amplification and next generation sequencing performance of single cancer cells. Oncotarget, 8(34), 56066–56080.
https://doi.org/10.18632/oncotarget.10701
4. Borgström, E. (2017). Comparison of whole genome amplification techniques for human single cell exome sequencing. PLOS ONE, 12(2), e0171566.
https://doi.org/10.1371/journal.pone.0171566
5. Normand, E. A. (2016). Comparison of three whole genome amplification methods for detection of genomic aberrations in single cells. Prenatal Diagnosis, 36(9), 823–830.
https://doi.org/10.1002/pd.4866
6. Raz, O. (2022). Whole-Genome Amplification—Surveying Yield, Reproducibility, and Heterozygous Balance, Reported by STR-Targeting MIPs. International Journal of
Molecular Sciences, 23(11), 6161.
https://doi.org/10.3390/ijms23116161









![[新品上市] ELISA PathRF – 有效消除類風濕因子和嗜異性抗體所造成的偽陽性訊號](https://biocommander.com/wp-content/uploads/2022/03/ELISA-PathRF-500x383.png)

![[新品上市]提高準確 TB 診斷,杜絕結核傳染](https://biocommander.com/wp-content/uploads/2021/12/TB-文章-500x383.png)
![[新品上市] MabTech 免疫試劑適用於多種實驗動物模型](https://biocommander.com/wp-content/uploads/2021/09/Mabtech_ELISpot_IFN-gamma-500x383.png)
![[新品上市] 世翔提供從研究到藥物/疫苗開發 COVID-19 的免疫試劑](https://biocommander.com/wp-content/uploads/2021/07/post-2455-01-500x383.png)
![[新品上市] KOKEN AteloCell® Honeycomb 能直接放進老鼠體內的3D培養細胞](https://biocommander.com/wp-content/uploads/2021/07/Cover-3d-culture-koken-honeycomb-500x383.png)
![[新品上市] TdB 提供葡聚醣和聚蔗糖衍生物 GPC 分析](https://biocommander.com/wp-content/uploads/2021/07/Cover-TdB-GPC-analysis-500x383.png)
![[新品上市] 3D 影像判讀於ELISPOT / FluoroSpot分析優勢](https://biocommander.com/wp-content/uploads/2021/07/post-2383-01-500x383.jpg)



![[新品上市] StemRNA Reprogramming system Introduction](https://biocommander.com/wp-content/uploads/2021/07/StemRNA-Reprogramming-system-Introduction-500x383.png)